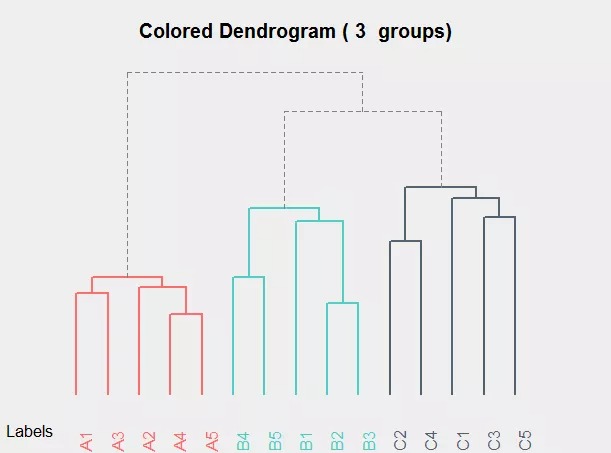
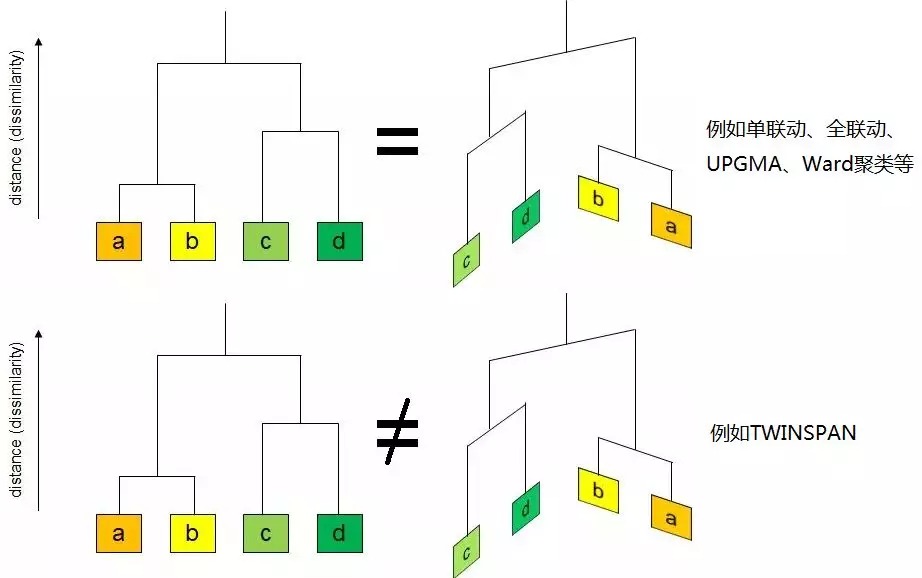
对于节点周围分支的方向,大多数层次聚类方法中都可以任意调整顺序;少数方法如TWINSPAN,对象的排列顺序和其分类特征密切相关,分支方向不可随意调整。
在前文简介层次聚合分类时,已经在R中展示了聚类树的一些简单调整方法,本篇继续作为延伸,展示一些更详细的可视化方案。
层次聚类
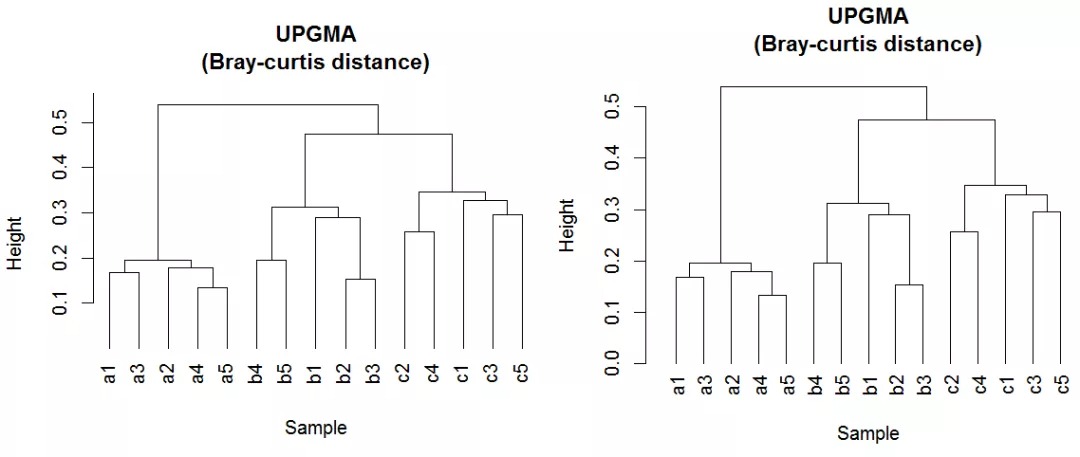
示例数据为来自16S测序所得的15个样本的细菌OTU丰度表,首先执行层次聚类识别样本归类。
#读取 OTU 丰度表
dat <- read.delim('otu_table.txt', row.names = 1, sep = '\t', head = TRUE, check.names = FALSE)
dat <- t(dat)
#样本分组文件
group <- read.delim('group.txt', row.names = 1, sep = '\t', head = TRUE, check.names = FALSE, stringsAsFactors = FALSE)
#计算样本间距离,以群落分析中常用的 Bray-curtis 距离为例
dis_bray <- vegan::vegdist(dat, method = 'bray')
#层次聚类,以 UPGMA 为例
upgma <- hclust(dis_bray, method = 'average')
upgma
plot(upgma, main = 'UPGMA\n(Bray-curtis distance)', sub = '', xlab = 'Sample', ylab = 'Height')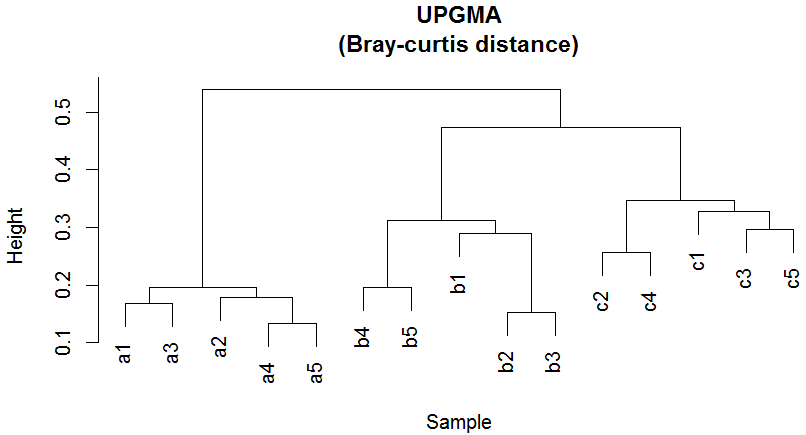
接下来,展示一些可能用到的聚类树调整方案。
注:下文所展示的方法仅为树状图本身的调整。其它组合类型的样式,如聚类树+柱形图、聚类树+热图、聚类树+排序图等,将放在后续的教程中绘制。
直接在plot()作图时添加参数调整
基本的参数调整已在层次聚合分类时提到,以下是继续延伸的内容。
#将样本高度保持在同一水平,以下两种方法都可以 par(mfrow = c(1, 2)) plot(upgma, hang = -1, main = 'UPGMA\n(Bray-curtis distance)', sub = '', xlab = 'Sample', ylab = 'Height') plot(as.dendrogram(upgma), main = 'UPGMA\n(Bray-curtis distance)', sub = '', xlab = 'Sample', ylab = 'Height')
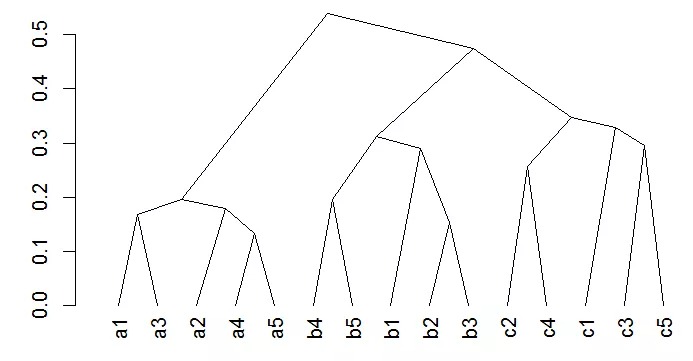
#三角形的聚类树 plot(as.dendrogram(upgma), type = 'triangle')
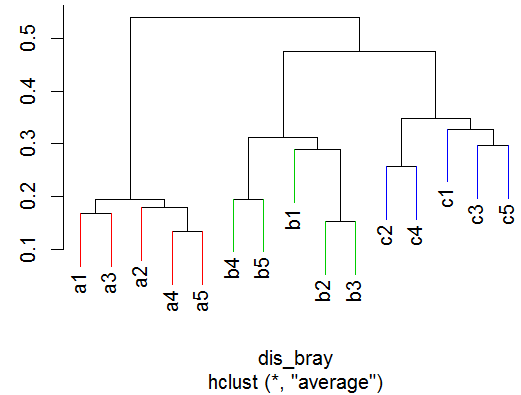
#给样本标记颜色,可以根据聚类后的簇进行标记,也可以根据先验分组标记
#这里按先验分组标记
clusMember <- group$cluster
names(clusMember) <- group$samples
labelColors <- c('red', 'blue', 'green3')
#标记颜色
colLab <- function(n) {
if (is.leaf(n)) {
a <- attributes(n)
labCol <- labelColors[clusMember[which(names(clusMember) == a$label)]]
attr(n, 'nodePar') <- c(a$nodePar, lab.col = labCol)
}
n
}
clusDendro <- dendrapply(as.dendrogram(upgma), colLab)
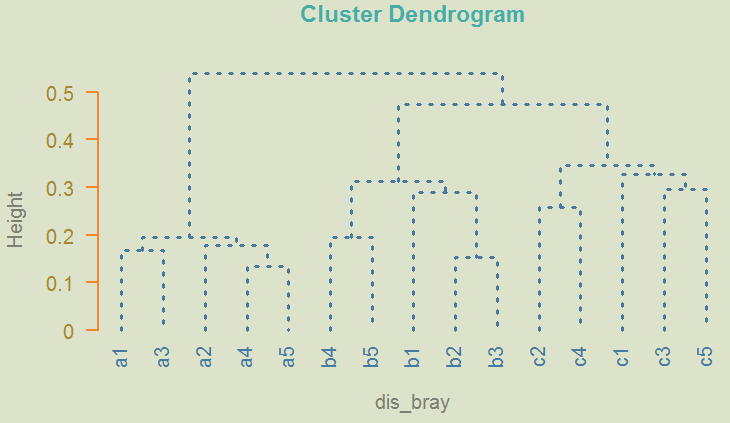
#聚类树
plot(clusDendro, main = 'UPGMA\n(Bray-curtis distance)', sub = '', xlab = 'Sample', ylab = 'Height')#配合 R 的其它基础作图函数使用,可自定义更改聚类树主题,例如 #绘制聚类树主体 op <- par(bg = '#DDE3CA') plot(upgma, col = '#487AA1', col.main = '#45ADA8', col.lab = '#7C8071', col.axis = '#F38630', lwd = 3, lty = 3, sub = '', hang = -1, axes = FALSE) #高度刻度轴 axis(side = 2, at = seq(0, 0.5, 0.1), col = '#F38630', labels = FALSE, lwd = 2) mtext(seq(0, 0.5, 0.1), side = 2, at = seq(0, 0.5, 0.1), line = 1, col = '#A38630', las = 2)
#用基础作图函数绘制进化树,github 上弄到的,又自己改了下,然后原出处找不到了......
treeplot <- function(tree, grp, grpcol, group_names, ...) {
treeline <- function(pos1, pos2, height, col1, col2)
{
meanpos = (pos1[1] + pos2[1]) / 2
segments(y0 = pos1[1] - 0.4, x0 = -pos1[2], y1 = pos1[1] - 0.4, x1 = -height, col = col1,lwd = 2)
segments(y0 = pos1[1] - 0.4, x0 = -height, y1 = meanpos - 0.4, x1 = -height, col = col1,lwd = 2)
segments(y0 = meanpos - 0.4, x0 = -height, y1 = pos2[1] - 0.4, x1 = -height, col = col2,lwd = 2)
segments(y0 = pos2[1] - 0.4, x0 = -height, y1 = pos2[1] - 0.4, x1 = -pos2[2], col = col2,lwd = 2)
}
plot(0, type = 'n', xaxt = 'n', yaxt = 'n', frame.plot = FALSE, xlab = '', ylab = '',
ylim = c(0, length(tree$order)),
xlim = c(-max(tree$height), 0))
legend('topleft',legend = group_names,pch = 15, col = grpcol, bty = 'n', cex = 1.5)
meanpos = matrix(rep(0, 2 * length(tree$order)), ncol = 2)
meancol = rep(0, length(tree$order))
for (step in 1:nrow(tree$merge))
{
if(tree$merge[step, 1] < 0){
pos1 <- c(which(tree$order == -tree$merge[step, 1]), 0)
col1 <- grpcol[as.character(grp[tree$labels[-tree$merge[step, 1]],1])]
}else {
pos1 <- meanpos[tree$merge[step, 1], ]
col1 <- meancol[tree$merge[step, 1]]
}
if(tree$merge[step, 2] < 0){
pos2 <- c(which(tree$order == -tree$merge[step, 2]), 0)
col2 <- grpcol[as.character(grp[tree$labels[-tree$merge[step, 2]],1])]
}else {
pos2 <- meanpos[tree$merge[step, 2], ]
col2 <- meancol[tree$merge[step, 2]]
}
height <- tree$height[step]
treeline(pos1, pos2, height, col1, col2)
meanpos[step, ] <- c((pos1[1] + pos2[1]) / 2, height)
if (col1 == col2){
meancol[step] <- col1
}else {
meancol[step] <- 'grey'
}
}
tree$order
for (y in tree$order) text(x = 0, y = y, labels = rownames(grp)[y], col = grpcol[grp[y,1]])
}
grpcol <- c('red', 'blue', 'green3')
names(grpcol) <- c('1', '2', '3')
treeplot(tree = upgma, grp = group[2], grpcol = grpcol, group_names = c('A', 'B', 'C'))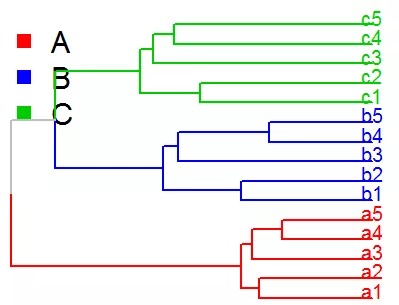
ape包中的系统发育树风格
聚类树和系统发育树都是树形图,因此也可以通过系统发育树的样式作图,例如ape包中的方法。
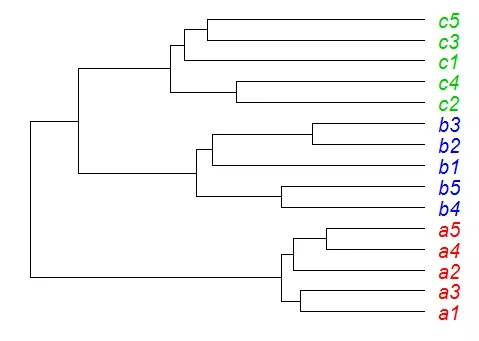
library(ape) #默认风格 plot(as.phylo(upgma))
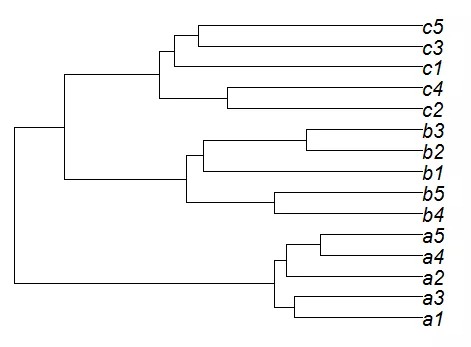
#“无根树”的聚类树样式 plot(as.phylo(upgma), type = 'unrooted')
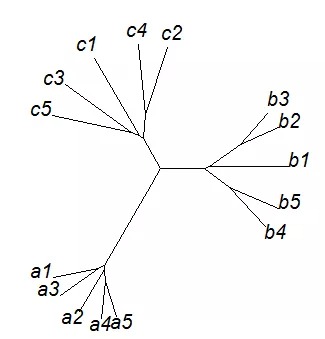
#环形的树 plot(as.phylo(upgma), type = 'fan')
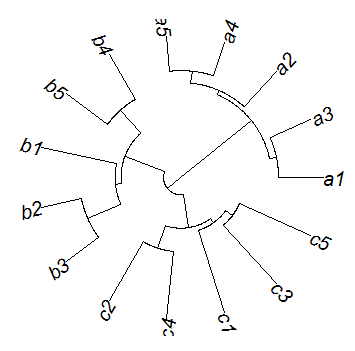
#给样本标记颜色,同上文,按样本已知的先验分组标记 plot(as.phylo(upgma), tip.color = labelColors[clusMember], label.offset = 0.01, cex = 1)
sparcl包的可视化
例如,给分支标记颜色。
#使用 sparcl 包给分支标记颜色 library(sparcl) ColorDendrogram(upgma, y = clusMember, labels = names(clusMember), branchlength = 0.1)
A2R包的可视化
一个彩色聚类树示例,脚本也可以下载下来自定义编辑。
# load code of A2R function
source('http://addictedtor.free.fr/packages/A2R/lastVersion/R/code.R')
# colored dendrogram
op <- par(bg = '#EFEFEF')
A2Rplot(upgma, k = 3, boxes = FALSE, col.up = 'gray50', col.down = c('#FF6B6B', '#4ECDC4', '#556270'))